

|
|
Seite 40 von 77 |






Oben in der Liste erscheinen perfekte Alignments mit E-Wert = 0 und Query cover = 100%: In der Datenbank wurden Sequenzen aufgespürt, die von Anfang der Abfragesequenz bis zu deren Ende mit ihr identisch sind.
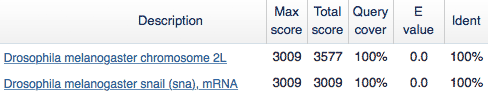
Beim ersten Eintrag handelt es sich um einen Abschnitt des Chromosomenarms 2L, der den snail-Locus einschließt. Er liefert uns das gleiche Alignment wie die snail mRNA. Im Prinzip können wir demnach nicht unterschieden, ob es sich bei der Abfragesequenz um die Sequenz einer vollständigen cDNA von snail handelt oder aber um eine Sequenz eines genomischen Fragments, denn das snail-Gen besitzt offenbar keine Introns.
Wenn man im Browser-Fenster noch weiter nach unten scrollt, kommt man zu den eigentlichen paarweisen Alignments.