

|
|
Seite 47 von 77 |






Bisher haben wir ausschließlich Nucleotide BLAST (blastn) verwendet und mit einer Nukleotid-Abfragesequenz die Datenbank von Nukleotidsequenzen durchsucht. Auf der BLAST-Einstiegsseite finden Sie eine Übersicht weiterer BLAST-Varianten, bei denen andere Kombinationen von Abfragesequenz und Datenbank verwendet werden.
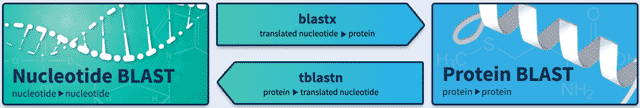
| blastn | vergleicht eine Nukleotidsequenz mit einer Nukleotiddatenbank |
| blastp | vergleicht eine Proteinsequenz mit einer Proteindatenbank (Protein BLAST) |
| blastx | vergleicht eine in allen sechs Leserastern (beide Stränge!) übersetzte Nukleotidsequenz mit der NCBI-Proteindatenbank |
| tblastn | vergleicht eine Proteinsequenz mit einer in allen sechs Leserastern übersetzten Nukleotiddatenbank |
| tblastx | vergleicht eine in allen sechs Leserastern übersetzte Nukleotidsequenz mit einer in allen sechs Leserastern übersetzte Nukleotiddatenbank |