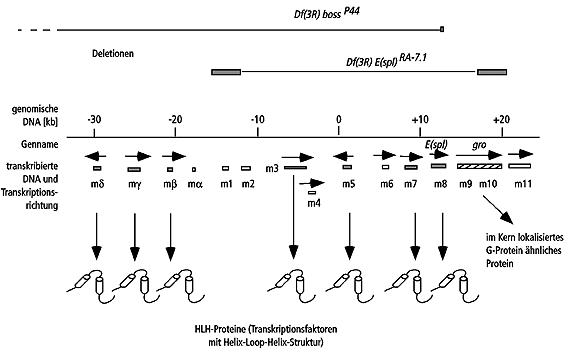
Abb. 34-15: Molekulare Karte des Enhancer-of-split-Genkomplexes. Die 7 schattierten Transkripte codieren für untereinander homologe Transkriptionsfaktoren des basic-Helix-Loop-Helix-Typs (bHLH) und stellen den eigentlichen Genkomplex dar. Da Punktmutationen
in diesen Transkriptionseinheiten nur schwach mutante Phänotypen erzeugen, müssen sich die unterschiedlichen Genprodukte in ihrer Funktion teilweise ersetzen können (Redundanz). Für den Komplex namensgebend ist das Enhancer-of-split[E(spl)]-Gen. Eine kleine
Defizienz in diesem Gen, die zu einer Kürzung des Helix-Loop-Helix-Proteins sowie zur Addition von 9 neuen Aminosäuren am Carboxyterminus führt, bewirken eine dominante Verstärkung des Effekts der Notchspl-Mutation auf die Augenentwicklung. Insgesamt 14
Transkriptionseinheiten produzieren 15 mRNAs (md, mg, mb, ma, m1-m11). Transkriptionseinheiten m9 bzw. m10 sind unterschiedlich terminierte mRNAs von groucho, die aufgrund ihrer unterschiedlichen Herkunft und Funktion nicht mehr zum Enhancer-of-split-Genkomplex
gezählt werden. Die Defizienz E(spl)RA-7.1 bewirkt einen starken neurogenenen Phänotyp, der jedoch von der Defizienz Df(3R) bossP44 noch übertroffen wird, was darauf hinweist, daß auch die außerhalb von Enhancer of splitRA-7.1 gelegenen Gene an neurogenen
Funktionen beteiligt sind.
 
|