

Seite 66 von 77 |






Bei der BLAST-Abfrage von Protein-Datenbanken, etwa mit blastx oder blastp, wird in den Scoring Parameters die verwendete Austauschmatrix festgelegt.
Standardmäßig ist die BLOSUM62-Matrix eingestellt, die für die meisten Zwecke sinnvolle Ergebnisse liefert.
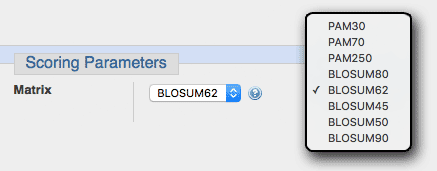
Hohe BLOSUM-Indexe zeigen an, dass die entsprechende Austauschmatrix auf der Basis sehr ähnlicher Sequenzen errechnet worden ist. BLOSUM80 bietet sich daher für die Suche nach Sequenzen an, die sich nur wenig von der Abfragesequenz unterscheiden.
Wenn noch Sequenzen geringerer Ähnlichkeit erfasst werden sollen, kann die BLOSUM45-Matrix eingesetzt werden.
Bei den PAM-Matrizes ist die Nomenklatur umgekehrt. Die PAM30-Matrix wurde für Sequenzen erstellt, bei denen ein geringerer Teil von Aminosäuren von einem Austausch betroffen ist als bei der PAM70-Matrix. PAM70 würde daher für den Vergleich entfernt-verwandter Sequenzen benutzt, PAM30 für stringentere Suchen.